Detección y cuantificación de virus dengue 2 en lisado celular y plasma de niños por qPCR en tiempo real usando un estuche comercial y el equipo EcoTM System-Illumina
Detection and quantification of dengue virus 2 in culture supernatant and plasma of children by qPCR using a commercial kit and the EcoTM System-Illumina device
Federico Perdomo Celis1, Piedad Perilla1, Doris M. Salgado1,2, Carlos F. Narváez1*
Resumen
Métodos para diagnóstico de dengue virus (DENV) en zonas endémicas son altamente necesarios. Uno de ellos es la reacción en cadena de polimerasa en tiempo real (qPCR), método que permite además la cuantificación del genoma viral. Los estuches comerciales de qPCR para DENV son costosos y restringen su uso a un número pequeño de dispositivos de qPCR, limitando la aplicación de la técnica. Aquí se evaluó el desempeño de un estuche comercial de qPCR para la detección de DENV-2 en un dispositivo de qPCR (EcoTM System, Illumina) localmente disponible, no listado por el fabricante del estuche. Lisado de células VERO-76 y plasma de niños, ambos con infección confirmada por DENV-2, fueron evaluados. Como controles de especificidad, lisado celular y plasma de niños infectados con DENV-1, además de lisado no infectado, fueron también incluidos. Las reacciones fueron además evaluadas simultáneamente en un equipo Applied Biosystem 7300, uno de los recomendados por el fabricante del estuche. La curva estándar generada por el EcoTM fue robusta (R2= 0.99), con baja variabilidad en las réplicas (<10%). La eficiencia de la reacción fue buena (88.8%) y sólo hubo amplificación en los lisados y plasma de niños infectados con DENV-2. Hubo una fuerte correlación positiva (R2= 1.0, P=0.0028) entre el número de copias de ARN viral en las muestras detectadas por los dos dispositivos de qPCR usados. Así, el uso del estuche para detección de DENV-2 aquí probado puede extenderse al EcoTM de forma segura. Este trabajo fortalece la capacidad tecnológica para el estudio de DENV en un área endémica.
Palabras clave: PCR en tiempo real, dengue, diagnóstico de dengue, estuche comercial, Dispositivo de PCR en tiempo real.
Abstract
Methods for Dengue virus (DENV) diagnosis in endemic areas are greatly needed. One of them is the real-time polymerase chain reaction (qPCR) that also enables to quantitate the viral genome. Kits of qPCR for DENV are expensive and restrict their use to a small number of qPCR devices, which limits the application of the technique. Here, we evaluated the performance of a commercial kit of qPCR to DENV-2 detection on a locally available qPCR device (EcoTM System, Illumina), not cited by the kit manufacturer. VERO-76 cells lysate and plasma from children, both with confirmed ongoing DENV-2 infection, were evaluated. As specificity control, cell lysates and plasma from children infected with DENV-1, and uninfected lysate, were also included. The reactions were simultaneously evaluated in an Applied Biosystems 7300 device. The standard curve generated by EcoTM was robust (R2= 0.99) with low variability in the replicates (<10%). The reaction efficiency was high (88.8%) and signal was only obtained in lysates and plasma infected with DENV-2. There was a strong positive correlation (R2= 1.0, P= 0.0028) between the number of copies of viral RNA in the samples detected by both qPCR devices. Thus, the use of the evaluate kit for detection of DENV-2 here tested can be extended to EcoTM. With this work, technological capacity for DENV study in an endemic zone is greatly strengthened.
Keywords: Real-time polymerase chain reaction, dengue, Diagnosis, commercial kits, Real-time PCR device.
Introducción
El dengue constituye la arbovirosis más importante a nivel mundial por su alta morbi-mortalidad e impacto económico y social. Se estima que 390 millones de infecciones ocurren anualmente en el mundo y debido a la localización del vector en áreas tropicales, más de 2.5 billones de personas están en riesgo de infectarse1. Colombia no es ajena a éste problema y en el último año se presentaron 61,649 casos de dengue, el 3.3% de ellos fueron formas severas de la infección. Localmente, el Departamento del Huila es un área hiperendémica para el virus, con 1,231 casos severos (17.3%) del total nacional en las recientes epidemias entre los años 2010 y 20122.
El dengue virus (DENV) pertenece al género Flavivirus, está compuesto de una membrana lipídica que envuelve la cápside y su genoma está conformado por ácido ribonucleico (ARN) de cadena sencilla y polaridad positiva3. Para el diagnóstico de la enfermedad se requieren criterios clínicos y de laboratorio. Varios métodos diagnósticos están disponibles para éste propósito y pueden ser clasificados como directos (detección de proteínas virales, amplificación de su genoma o aislamiento viral) o indirectos (detección de anticuerpos específicos para DENV)4. Para la detección del genoma viral comúnmente es utilizada la reacción en cadena de polimerasa por transcripción inversa (RT-PCR)5. Esta técnica se basa en la transcripción del ARN a ácido desoxirribonucleico (ADN) utilizando transcriptasas inversas, para posteriormente realizar la amplificación del segmento de interés gracias a polimerasas dependientes de ADN. La reacción puede llevarse a cabo en uno o dos pasos (la síntesis del ADNc y su amplificación se hacen en un mismo tubo, o por separado). Además puede ser anidada o semianidada, es decir, primero se realiza la reacción con primers externos para amplificar una región de ADN extensa que contiene el segmento de interés y luego utilizar éste producto como molde para una segunda PCR con uno (semianidada) o dos (anidada) primers internos para amplificar la región específica6.
Una variación de ésta es la PCR en tiempo real (qPCR), que tiene como característica fundamental la posibilidad de determinar el número de copias de ARN viral7. En la qPCR se introducen moléculas fluorescentes que se unen a los ácidos nucleicos y permiten detectar la amplificación del segmento de interés al tiempo en que van ocurriendo los ciclos de la reacción, calculando finalmente la cantidad relativa (veces de incremento en la amplificación del gen de interés en comparación con la de un gen de concentración conocida) o absoluta (determinación a través de una curva estándar generada por la dilución seriada de un control positivo) del genoma viral8. Se ha propuesto que la qPCR tiene ventajas sobre la PCR convencional, como por ejemplo el ser más sensible que ésta9.
Existen diversos protocolos de detección, serotipificación y cuantificación de DENV por qPCR. Su diferencia se basa principalmente en la química fluorescente utilizada, si se utilizan reacciones en uno o dos pasos o si se detectan uno o más serotipos al mismo tiempo (qPCR en formato multiplex). Las moléculas fluorescentes de detección de la amplificación más frecuentemente utilizados son los intercaladores de ADN de cadena doble, como SYBR Green10, 11 y las sondas de hidrólisis fluorescentes, como Taqman9, 12, 13. Los primeros emiten fluorescencia cuando se unen a ADN de cadena doble, mientras que los segundos lo hacen cuando la molécula de detección se separa de otra que inhibe su fluorescencia7. En el caso de las reacciones en uno o dos pasos, se prefieren los primeros porque se minimiza la probabilidad de contaminación de la muestra y la variación entre las reacciones enzimáticas14, 15. Como característica, la mayoría de éstos emplean sondas de hidrólisis tipo Taqman16, 17.
Actualmente para el diagnóstico de dengue en humanos existe sólo aprobado un estuche de qPCR desarrollado por el centro para el control y prevención de enfermedades (CDC) y aprobado por la FDA (del inglés Food and Drugs Administration) de Estados Unidos18 y se ha demostrado que tiene alta sensibilidad y especificidad19. Éste permite la detección de los cuatro serotipos virales en muestras de plasma o suero a través de una reacción en un sólo paso utilizando sondas de hidrólisis tipo Taqman, siendo el ABI 7500 Fast Dx (Applied Biosystem) el único equipo recomendado para la aplicación del estuche. Sin embargo, en distintos laboratorios se han desarrollado ensayos de qPCR para la detección de DENV con fines investigativos20, 21 y además existen varios estuches comerciales con el mismo propósito que han demostrado un buen rendimiento22. Una limitación importante para el uso de éstos últimos es su alto costo y el bajo número de equipos de qPCR recomendados por cada uno de ellos para llevar a cabo el ensayo, la mayoría de equipos no disponibles en regiones alejadas y endémicas. Esto limita enormemente la aplicación de la técnica y el posible impacto en el diagnóstico rápido de la infección. Aquí, se realizó la detección y cuantificación de DENV-2 por qPCR en lisado celular y plasma de niños infectados usando el estuche comercial de detección Dengue Virus II Real Time RT-PCR Kit (Shanghai ZJ Bio-Tech Co) aplicado en el equipo de qPCR EcoTM System (Illumina, San Diego, CA), que no ha sido listado entre los dispositivos recomendados para la aplicación del respectivo estuche comercial, pero que está localmente disponible. Además, se comparó simultáneamente con un segundo equipo recomendado por los fabricantes del estuche.
Materiales y métodos
Pacientes y muestras
Dos niños agudamente infectados con dengue fueron incluidos en éste estudio. Para el diagnóstico de dengue se emplearon los criterios revisados de la OMS 2009 presentes en la guía integrada de pacientes con dengue, Colombia 2010. Previo consentimiento y asentimiento informado, dos a cuatro mililitros de sangre total fueron tomados de cada niño en tubos con EDTA, ambos casos en la etapa aguda de la infección (primeros 5 días de iniciada la fiebre). La sangre fue centrifugada a 300 x g, el plasma separado, alicuotado y conservado a -70°C hasta su uso para detección del antígeno NS1 viral y de las inmunoglobulinas (Ig) M, G específicas para DENV (Panbio, Alere, Australia) y la RT-PCR convencional.
Elisa para diagnóstico de dengue
Para la detección en plasma de IgM e IgG específicas de DENV y la proteína NS1 viral se emplearon los estuches comerciales Dengue IgM capture Elisa (Ref: E-DEN01M), Dengue IgG capture Elisa (Ref: E-DEN02G) y Dengue Early Elisa (Ref: E-DEN02P), respectivamente (todos de Panbio, Alere, Australia), siguiendo todas las instrucciones del fabricante. Brevemente, para la detección de las Igs, 100μL de las muestras, controles y calibrador diluidos 1/100 en el diluyente proveído por el estuche, fueron añadidos a la placa cubierta con anticuerpo anti-IgM o IgG humanas. Luego de una hora de incubación a 37°C, se lavó la placa y se añadieron 100μL del antígeno viral previamente mezclado con el anticuerpo anti-dengue conjugado a peroxidasa. Se incubó nuevamente por una hora a 37°C y luego de un lavado, se reveló con 100μL de tetrametilbenzidina (TMB). La reacción fue detenida con 100μL de ácido fosfórico 1M. La lectura se realizó a 450 nm (referencia 600-650 nm) en un equipo ELx 800 (BIO-TEK Instruments, Winooski, Estados Unidos). En la detección de la proteína NS1, 100μL de las muestras, controles y calibrador diluidos al medio fueron añadidos a la placa cubierta con el anticuerpo anti-NS1. Luego de una hora de incubación a 37°C la placa se lavó y se añadieron 100μL del anticuerpo anti-NS1 conjugado a peroxidasa. Se incubó nuevamente la placa por una hora a 37°C, y luego de otro paso de lavado, se reveló y se leyó como se describió previamente.
Amplificación de DENV-1 y 2 en cultivo celular
DENV-1 y 2 fueron amplificados en cultivo de células VERO-76 (adquiridas desde el ATCC, Número: CRL-1587D). Quinientos μL de sobrenadante de cultivo con DENV-1 y 2 (amablemente proveídos por Iván Darío Vélez, Universidad de Antioquia, Colombia) fueron añadidos a una monocapa de células VERO-76 con un 70% de confluencia. Éstas se incubaron en medio DMEM 1X y SFB al 2% (ambos de GIBCO, Carlsbad, Estados Unidos; Cat: 11965-126 y 16000-044, respectivamente) a 37ºC y 5% de CO2. Al 5 día de cultivo se verificó la aparición de efecto citopático sobre la monocapa por microscopía óptica invertida y posteriormente el sobrenadante de los cultivos se recolectó, se centrifugó y se congeló a -70°C hasta su uso. Como control, se recolectó el sobrenadante de cultivo celular no infectado tratado de forma idéntica a la ya descrita. La pureza del serotipo de DENV infectante fue determinada por RT-PCR convencional realizada como se describe abajo.
Aislamiento del ARN para la RT-PCR y qPCR
Para la purificación del ARN se utilizó el estuche comercial QIAmp viral RNA (QIAGEN, Valencia, CA, Cat: 52906), siguiendo todas las recomendaciones del fabricante. Brevemente, 140μL de las muestras fueron mezclados con buffer AVL y “carrier” de ARN e incubados a temperatura ambiente por 10 min. Luego se añadieron 560μL de etanol al 100% y tras 2 pasos de centrifugación a 6,000 xg por un min se pasaron 1,260μL de la solución total por la columna correspondiente. Posteriormente se realizaron 2 lavados con 500μL de buffer AW1 y AW2 y se hizo la elución con 40μL de buffer AVE. La concentración de ARN aislado de cada condición fue determinada usando el equipo NanoDrop 2000 (Thermo, Wilmington, Estados Unidos), obteniendose una mediana de 98.7 ng/μL (rango 91-119.7).
Detección y tipificación de DENV en lisado y plasma por RT-PCR convencional
El serotipo infectante en las muestras analizadas fue determinado por RT-PCR convencional como previamente ha sido reportado5, 23. 5μL del ARN previamente aislado fueron añadidos a una mezcla que contenía KCl 50 mM, Tris 10 mM (pH 8.5), Triton X-100 0.1%, gelatina 0.01%, cada uno de los cuatro desoxinucleótidos trifosfato (DNTPs) a una concentración de 2.5 mM, MgCl2 1.5 mM, ditiotreitol 5 mM, primers externos e internos (serotipo-específicos) a una concentración de 1 μg/μL y 5 U/μL de Taq ADN polimerasa (Promega Corp., Madison, Wis) Las secuencias de los primers serotipo-específicos usados fueron las siguientes: DENV- 1: 5’-TCA ATA TGC TGA AAC GCG CGA GAA ACC G-3’; DENV-2: 5’-TTG CAC CAA CAG TCA ATG TCT TCA GGT TC-3’; TS1: 5’-CGT CTC AGT TGA TCC GGG GG-3’; TS2: 5’- CGC CAC AAG GGC CAT GAA CAG-3’. La transcripción inversa y la amplificación se llevaron a cabo en un termociclador VeritiTM (Applied Biosystem). El perfil térmico usado fue: 42°C por 60 minutos, seguido de 35 ciclos de amplificación a 94°C por 45 segundos, 60°C por 45 segundos y 72°C por 1 minuto, con una extensión final a 72°C por 7 minutos.
PCR en Tiempo Real
Para la amplificación y cuantificación de DENV-2 se empleó un estuche comercial (Shanghai ZJ Bio-Tech Co, Shanghai, China, Cat: ER-0063-04), que emplea para la detección una sonda fluorescente tipo Taqman. Se siguieron todas las instrucciones del fabricante. Brevemente, 9μL de Super Mix, 0.5μL del mix enzimático y 0.5μL del control interno (para detectar la inhibición de la PCR) fueron mezclados con 2.5μL del control positivo (cuatro diluciones seriadas 1:10, iniciando en 1X107 copias/mL), del control negativo (agua libre de nucleasas) o del ARN extraído de las muestras. Debido a la capacidad de los pozos del dispositivo de qPCR utilizado, el volumen final de la reacción se ajustó a 12.5μL y no 25μL como recomienda el estuche comercial. Cada muestra, excepto el control negativo, se analizó por duplicado. El perfil térmico que se empleó fue: 45°C por 10 minutos, 1 ciclo; 95°C por 15 minutos, 1 ciclo; 95°C por 15 segundos y 60°C por 60 segundos, 40 ciclos; la fluorescencia fue medida a 60°C. La reacción se llevó a cabo en el equipo EcoTM System (Illumina, San Diego, CA). Los datos fueron analizados con el software proveído por el equipo (EcoStudy software versión 5.0). La sensibilidad y el rango de detección del ensayo fueron 1X103 copias/mL y 2X103-1X108 copias/mL, respectivamente24. Se tomó como cut off un Cq (ciclo en el que cada muestra sobrepasó el umbral de detección) de 37 ciclos.
Análisis Estadístico
El análisis estadístico se hizo con el software GraphPad Prism 5.0. Para hallar el grado de correlación entre variables se utilizó el coeficiente de determinación, tomando como significativo un valor de P<0.05. Para propósitos estadísticos, a las muestras con número no detectable de copias se les asignó un valor arbitrario de 500 copias/ mL (la mitad del límite de sensibilidad del ensayo).
Resultados
Pacientes incluidos
Se incluyeron dos niños de 13 y 14 años, uno de sexo masculino y otro femenino, que ingresaron al Departamento de Pediatría del Hospital Universitario de Neiva. Ambos consultaron en los primeros 5 días de iniciada la fiebre. Los dos casos correspondieron clínicamente a dengue grave. El diagnóstico y tipo de infección se confirmó a través de la detección de NS1, IgM e IgG específicas en plasma. Ambos pacientes presentaron infección secundaria (relación IgM/IgG virus específicas <1.8). En la Tabla 1 se describen los resultados de las pruebas de Elisa realizadas para el diagnóstico de los pacientes.
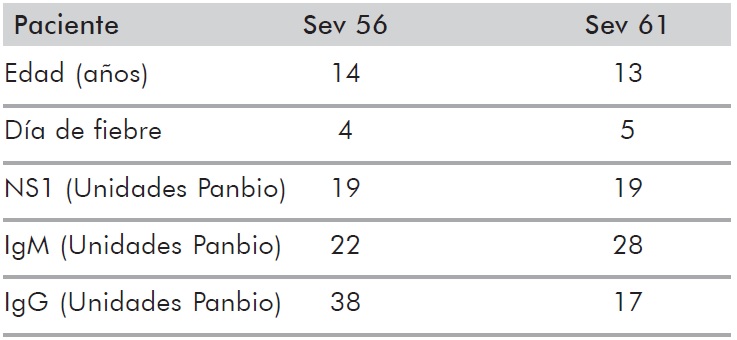
| Tabla 1. Resultados de las pruebas de Elisa realizadas para el diagnóstico de los pacientes | ||
| Paciente | Sev 56 | Sev 61 |
| Edad (años) | 14 | 13 |
| Día de fiebre | 4 | 5 |
| NS1 (Unidades Panbio) | 19 | 19 |
| IgM (Unidades Panbio) | 22 | 28 |
| IgG (Unidades Panbio) | 38 | 17 |
DENV-1 y 2 fueron los virus presentes en lisado celular y plasma, detectados por RT-PCR convencional
La pureza del serotipo amplificado por cultivo en células VERO- 76 fue determinada por RT-PCR convencional (Figura 1). Para ambos casos se emplearon controles positivos (lisados celulares, previamente tipificados) de cada uno de los serotipos virales. Con el protocolo usado se amplifican segmentos de la proteína preM de DENV-1 y 4 y de la proteína C (de la cápside) de DENV-2 y 3. Como ha sido previamente descrito y como es mostrado en la Figura 1, los productos de amplificación de DENV-1, 2, 3 y 4 generaron bandas de 482, 119, 290 y 389 pb, respectivamente23. La tipificación del DENV en el plasma de los niños mostró que los serotipos infectantes fueron DENV-1 y 2 (Figura 1B). Todas las muestras fueron tipificadas por lo menos dos veces, obteniendo similar resultado en cada análisis. Así, DENV-1 y DENV-2 fueron los serotipos infectantes detectados tanto en lisado celular (Figura 1A) como en plasma de niños con la forma severa de la enfermedad (Figura 1B).
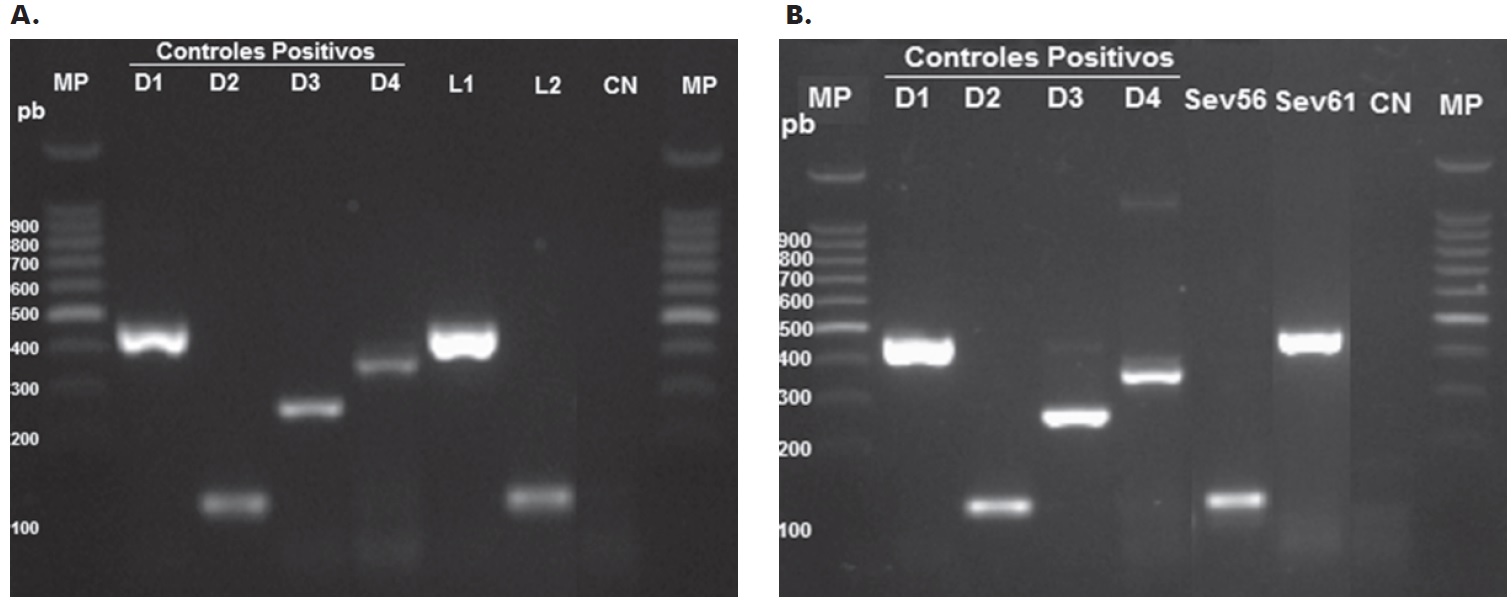

Figura 1. Serotipificación de las muestras de lisado celular (A) y plasma de niños (B) infectados con DENV por RTPCR convencional. MP: Marcador de peso; D1: Virus Dengue 1; D2: Virus Dengue 2; D3: Virus Dengue 3; D4: Virus Dengue 4; Sev 61 y Sev 56: Plasma de niños con infección por DENV; L1 y L2: Lisados celulares infectados con DENV- 1 y 2, respectivamente; CN: Control negativo.
Detección de DENV-2 en lisado y plasma por qPCR
Para la detección y cuantificación específica de DENV-2, ARN purificado de cada condición fue analizado usando un estuche comercial aplicado en el equipo EcoTM. Para conocer el número absoluto de copias de genoma viral/mL, se realizó la curva estándar proveída por el estuche que consta de cuatro diluciones seriadas del control positivo. Como muestra la Figura 2A, la curva estándar mostró un coeficiente de determinación del 99% y la amplificación tuvo una eficiencia del 88.8%. Este resultado demuestra que con el EcoTM se puede detectar y cuantificar DENV-2 de forma eficiente con el estuche empleado.
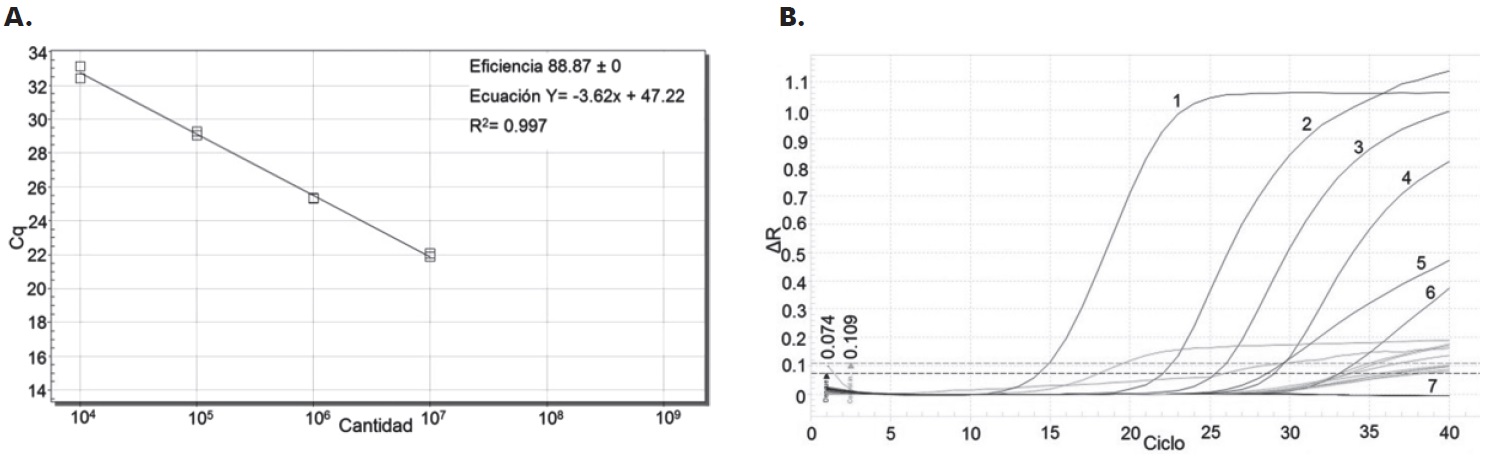

Figura 2. A. Curva estándar del estuche generada por el equipo EcoTM System, Illumina. En el eje Y se encuentra el ciclo de cuantificación (Cq), o ciclo en el que la señal de fluorescencia cruzó el umbral de detección, y en el eje X se encuentra la cantidad del gen presente en cada muestra (escala logarítmica). Cada cuadro representa un punto de la curva, analizados por duplicado (diluciones seriadas 1:10, partiendo desde 1X107 copias/mL hasta 1X104 copias/mL). En la esquina superior derecha se muestran la eficiencia de la amplificación en cada ciclo de la qPCR, la ecuación de la curva y el R2 de ésta. B. Amplificación linear (ΔR; unidades de fluorescencia normalizada al ruido de fondo en cada pozo) de las muestra a través de los ciclos de PCR en EcoTM. Las líneas a trazos cortos azul y verde representan el umbral de detección para las muestras (0,074) y para el control interno de inhibición de la PCR (0,109), respectivamente; todas las muestras que sobrepasaron éste umbral fueron cuantificadas. Cada línea azul representa a una muestra o estándar y las líneas verdes representan sus respectivos controles internos de amplificación. 1: Lisado celular infectado con DENV-2; 2: Control positivo, 1X107 copias/mL; 3: Control positivo, 1X106 copias/mL; 4: Control positivo, 1X105 copias/mL; 5: Plasma de niño con infección por DENV-2; 6: Control positivo, 1X104 copias/mL: 7: Lisado celular no infectado, lisado celular infectado con DENV-1 y plasma de niño con infección por DENV-1.
Usando la curva estándar y el Cq, de forma automatizada se determinó el número de copias de genoma viral de DENV-2 por mililitro en cada una de las muestras. Como se observa en la Figura 2B, muestras con infección por DENV-2 previamente confirmada por RT-PCR convencional tuvieron altas concentraciones de ARN viral, con un Cq que fue mucho menor a 37 (mediana 21, rango 14- 28). De notar, en el caso del lisado celular infectado por éste serotipo, la concentración de ARN viral fue incluso mayor al punto más alto de la curva estándar. En contraste, muestras de lisado celular y plasma con infección por DENV-1 o el mock, tuvieron un Cq >37 (mediana 40) y la concentración de ARN viral fue menor al límite de detección del ensayo (<1X103 copias/mL) (Tabla 2). También, como se observa en la Tabla 2, la diferencia del valor entre los duplicados fue menor al 10%, demostrando la baja variabilidad del ensayo.
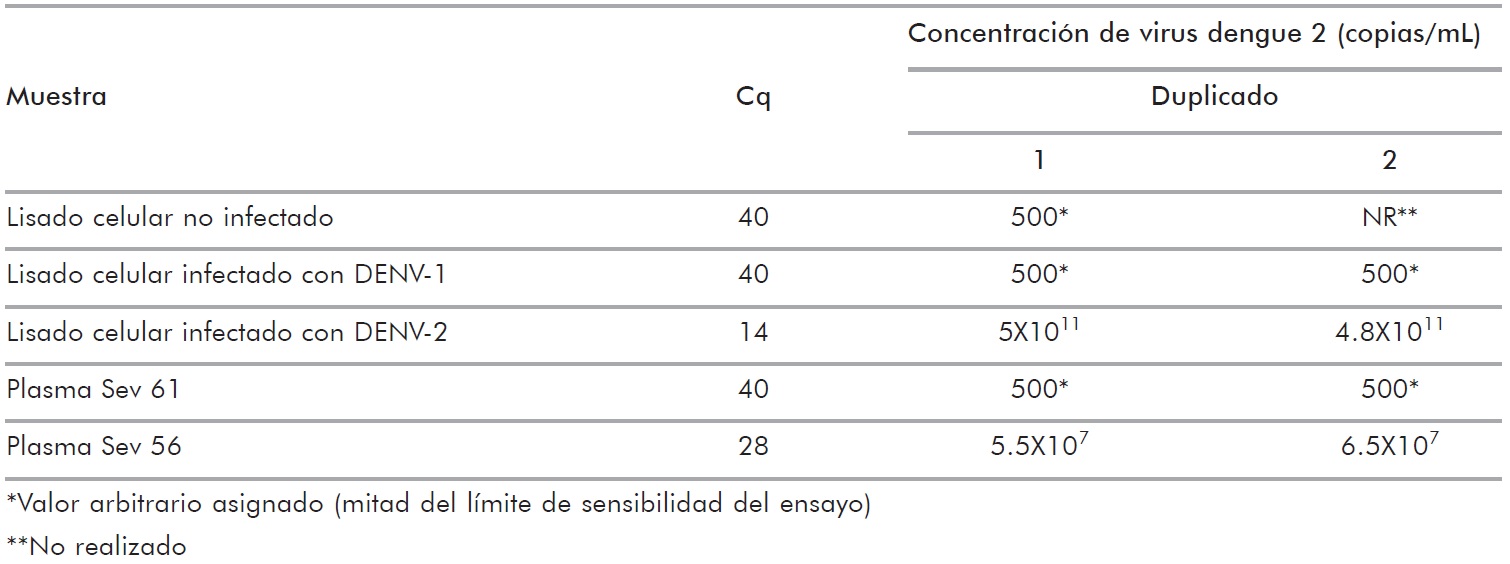
| Tabla 2. Número de copias de ARN de virus dengue 2 en las muestras, analizadas por duplicado | |||
| Concentración de virus dengue 2 (copias/mL) | |||
| Muestra | Cq | Duplicado | |
| 1 | 2 | ||
| Lisado celular no infectado | 40 | 500* | NR** |
| Lisado celular infectado con DENV-1 | 40 | 500* | 500* |
| Lisado celular infectado con DENV-2 | 14 | 5X1011 | 4.8X1011 |
| Plasma Sev 61 | 40 | 500* | 500* |
| Plasma Sev 56 | 28 | 5.5X107 | 6.5X107 |
| *Valor arbitrario asignado (mitad del límite de sensibilidad del ensayo) | |||
| **No realizado | |||
El rendimiento del EcoTM y el Applied Biosystem 7300 para la detección de DENV-2 usando el estuche de Shanghai ZJ Bio-Tech Co es comparable
Debido a que el equipo EcoTM no es listado entre los dispositivos recomendados para la cuantificación de DENV-2 con el estuche comercial de Shanghai ZJ Bio-Tech Co, se comparó el desempeño de éste con uno de los dispositivos recomendados por el mismo, el Applied Biosystem (ABI) 7300 (Applied Biosystem, Foster City, CA). La amplificación de las muestras se realizó simultáneamente en ambos equipos bajo las mismas condiciones previamente descritas (cada muestra por duplicado). Como es mostrado en la Figura 3, una fuerte correlación positiva fue encontrada entre el número de copias por mililitro de genoma de DENV detectados por los dos equipos. En resumen, a pesar de que el EcoTM no es recomendado por los fabricantes del estuche comercial Dengue Virus II Real Time RT-PCR Kit (Shanghai ZJ Bio-Tech Co) para la aplicación del ensayo, su desempeño es comparable al del equipo ABI 7300.
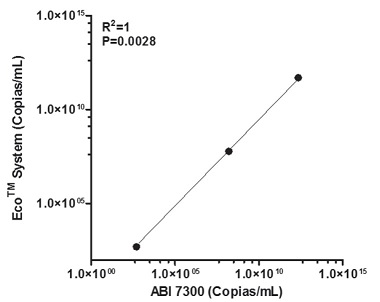

Figura 1. Correlación entre el número de copias/mL de DENV-2 detectado por los dos equipos de qPCR. El promedio del duplicado de cada muestra, analizadas en los equipos EcoTM y ABI 7300, fue comparado para hallar la correlación entre el número de copias/mL de ARN de DENV-2. Cada punto representa el promedio del duplicado de una muestra. En el extremo superior izquierdo se muestra el grado de correlación.
Discusión
Por qPCR, usando un estuche comercial y el equipo EcoTM se realizó la detección y cuantificación de DENV-2 en lisado celular y plasma de niños con infección natural por DENV, previamente serotipificados por RT-PCR convencional. En ambos tipos de muestras se detectó DENV-2 de forma específica y con un desempeño eficiente.
Se incluyeron dos muestras de plasma de niños con infección aguda por DENV diagnosticada y confirmada por la detección en plasma del antígeno viral NS1, IgM e IgG específicas para el virus. Ambas muestras fueron tomadas en los primeros 5 días de iniciada la fiebre y el serotipo infectante se determinó por RT-PCR convencional. Igualmente fueron analizados lisados celulares obtenidos de la infección de células VERO-76 con DENV-1 y 2. En los últimos años se han descrito diversos protocolos y estuches comerciales de detección y cuantificación de DENV en fluidos biológicos o en sobrenadantes de cultivos o lisados celulares, todos con fines investigativos. La mayoría de ellos son costosos y poco accesibles, además de estar restringidos a un número limitado de dispositivos de qPCR. Ya que localmente se dispone del equipo EcoTM, se evaluó su utilidad en la detección de DENV-2 (un serotipo viral altamente circulante en la región) con un estuche comercial, ya que éste último no lo lista dentro de los dispositivos para realizar el ensayo. Como química fluorescente, el estuche contiene sondas tipo Taqman, utilizadas en la mayoría de protocolos descritos para el análisis de DENV25. Ésta proporciona una menor tasa de falsos positivos en comparación con SYBR Green y evita tener que realizar análisis de curvas de “melt” para la detección de dímeros de primers9. Adicionalmente el ensayo incluye como control interno el fluorocromo VIC/JOE, que, además de permitirnos identificar una posible inhibición de la PCR (que puede ser causada por una mala calidad de la muestra o una alta concentración del ARN o ADN), también sirve como señal de referencia para que las variaciones en la fluorescencia de cada pozo (causada por ejemplo por errores en el pipeteo) sean normalizadas automáticamente en relación al ruido de fondo de la reacción por el dispositivo de qPCR, por lo tanto, la fluorescencia de un pozo no es afectada por la del pozo adyacente.
El número absoluto de copias de DENV-2 fue determinada a través de la realización de una curva estándar del control positivo incluido en el estuche (cuantificación absoluta)26. Sin duda, la cuantificación absoluta se prefiere sobre la relativa pues determina la cantidad exacta de copias, a pesar de la limitación de tener que contar con una concentración conocida del gen de interés27. La curva estándar obtenida en el ensayo tuvo una pendiente de -3.62, con una eficiencia de la PCR del 88.8%, es decir que la amplificación de las hebras molde de ADNc fue cercana al doble en cada ciclo de la PCR; de igual manera, hubo una correlación del 99.7% entre el Cq y las concentraciones de ADNc de la muestra de la curva estándar (Figura 2A). Estos valores son importantes para la confiabilidad de los cálculos de la cuantificación absoluta, pues son una medida de la optimización de la PCR a lo largo de los ciclos7. En general se acepta que la curva estándar debe tener una pendiente entre -3.68 y -3.10, con una eficiencia mayor del 85% y un coeficiente de correlación mayor del 90%11, 25. Todos los Cq de las muestras positivas para DENV-2 estuvieron por debajo de 37, valor que el CDC recomienda como cut off en éste tipo de ensayos por las bajas probabilidades de que se presenten falsos positivos con este valor19. Las muestras con infección por DENV-1 confirmada por RT-PCR convencional tuvieron Cq mayores a 37 y por lo tanto se consideraron negativas para el serotipo 2 (Figura 2B, Tabla 2). Debido a que en nuestra región más del 90% de los casos de dengue son producidos por DENV-1 y 2 (Narváez CF, resultados no publicados), era crítico determinar si el ensayo podría diferenciar de forma segura y específica estos dos serotipos. El estuche empleado sólo detectó DENV-2 en muestras con infección por éste serotipo, confirmada por RT-PCR convencional, y no en las infectadas por DENV-1, demostrando la especificidad del ensayo y su utilidad en regiones con alta circulación de estos dos serotipos.
Tanto los antígenos (principalmente NS1) como el ARN del DENV se encuentran elevados durante los primeros días de la enfermedad y es en este tiempo donde se debe hacer la detección y diagnóstico molecular19. Las muestras de plasma analizadas en el estudio fueron tomadas antes del 5 día de fiebre, encontrando una alta viremia en el paciente con infección por DENV-2 (media 6X107 copias/mL). Esto es consistente con reportes previos que muestran que durante los días 3 a 6 de iniciados los síntomas en la infección por DENV-2 la viremia alcanza niveles entre 106 a 108 copias/mL28, 29.
La mayoría de los estuches comerciales de qPCR recomiendan ciertos equipos para su realización. Ya que el EcoTM no se encuentra listado entre los dispositivos que soportan la detección de DENV- 2 por el estuche comercial de Shanghai ZJ Bio-Tech Co, se comparó su rendimiento con el obtenido con el dispositivo ABI 7300, que sí es listado por el inserto del estuche. Se obtuvo una fuerte correlación positiva entre los resultados obtenidos con ambos equipos. En resumen, a pesar de las pocas muestras biológicas analizadas, la confiabilidad de los resultados obtenidos se basa en i. El uso de lisado celular y plasma de niños naturalmente infectados con DENV- 2 (Figura 1); ii. La introducción de controles externos como muestras infectadas con un serotipo diferente a la especificidad ofrecida por el estuche; iii. El adecuado funcionamiento de los controles internos y externos; iv. La utilización de un segundo dispositivo de amplificación (Figura 3).
Debido a la importancia global del dengue y la necesidad de mejorar la capacidad diagnóstica e investigativa en áreas hiperendémicas, en varios laboratorios se han desarrollado ensayos de qPCR y en algunos casos se ha comparado su rendimiento tanto con protocolos de RT-PCR convencional21, como con estuches comerciales y de referencia (diseñado por el CDC)30. Sin embargo, a pesar de que éstos muestran buena funcionalidad, es difícil trasladar su aplicación a otros laboratorios debido a diferencias en los reactivos, dispositivos de qPCR o experiencia de los operadores. Los estuches comerciales representan una alternativa a este problema, pues garantizan la confiabilidad y reproducibilidad de los resultados22, pero tienen limitaciones como el alto costo y la restricción de la aplicación del ensayo en dispositivos de qPCR no recomendados por el fabricante, al igual que el estuche de referencia del CDC31. Por todo lo anterior, se hace necesario no sólo el diseño de nuevos ensayos de qPCR para la detección de DENV, sino también la evaluación de los ya disponibles para determinar su desempeño en dispositivos de qPCR con los que se disponga localmente32.
En conclusión, los resultados demuestran el adecuado desempeño del EcoTM en la detección y cuantificación de DENV-2 en plasma y lisado celular usando un estuche comercial, por lo que la implementación de ésta técnica servirá para impulsar grandemente estudios virológicos, inmunes y moleculares a nivel regional.
Agradecimientos
A Frank Barreiro, Laboratorio de Medicina Genómica, por la adquisición y análisis de los datos en el equipo ABI 7300. Éste trabajo fue financiado por la Vicerrectoría de Investigación y Proyección Social de la Universidad Surcolombiana.
